
Hiro Clinic 的检测精度(亲权肯定率)目标是:
99.99999999999%以上为目标。
假设地球上的人口为80亿人,出现错误的可能性为0.008人。这个结果是通过一种称为似然比的统计学指标计算出来的。借助STR(短串联重复序列)分析技术,有可能实现这一精度。
尤度比与亲权肯定率的关系
| 似然比 | 亲权肯定率 | 出错概率 |
|---|---|---|
| 10 | 90%以上 | 每10人中约有1人可能错误 |
| 1,000(3个0) | 99.9%以上(3个9) | 每1,000人中约有1人可能错误 |
| 100,000(5个0) | 99.999%以上(5个9) | 每10万人中约有1人可能错误 |
| 10,000,000(7个0) | 99.99999%以上(7个9) | 每1,000万人中约有1人可能错误 |
| 1,000,000,000(9个0) | 99.9999999%以上(9个9) | 每10亿人中约有1人可能错误 |
| 100,000,000,000(11个0) | 99.999999999%以上(11个9) | 每1,000亿人中约有1人可能错误 |
| 10,000,000,000,000(13个0) | 99.99999999999%以上(13个9) | 每10兆人中约有1人可能错误 |
由于 STR(短串联重复序列)在数值分布上具有更大的变异范围,因此能够实现更高精度的鉴定。
若仅使用 SNP(单核苷酸多态性)进行检测,则需超过 5,000 个不同的 SNP 位点,才能达到同等的准确度。
DNA亲子鉴定技术的现代发展
ABO血型系统于1900年被发现,并在20世纪30年代开始用于亲子鉴定中的排除测试。随后,随着Rh血型抗原、MNS血型抗原以及人类白细胞抗原(HLA)等血液标记的引入,检测的准确性有所提升,但仍无法完全消除误差。
1977年,随着桑格法(Sanger sequencing)DNA测序技术的出现,使DNA检测成为可能,从而在亲子鉴定中实现了远超以往方法的统计学准确率。如今,DNA检测广泛应用于亲子关系鉴定、法医学调查、产前检测等多个领域,成为现代生物技术的重要支柱之一。
检测流程
若在诉讼程序中使用检测结果,相关人员的样本将由医生在诊所内采集。除此之外,您也可以使用简单易用的采样套件在家中自行采集样本并寄送。
样本在接收后将被登记至实验室数据库。随后,从采集的细胞中提取并纯化细胞核内的DNA。该流程通过高通量、全自动化系统完成,相较于传统DNA检测方法,具有更短的处理时间、更低的成本以及更高的准确性。
此外,本检测不对全基因组进行解析,而是采用以特定标记对立等位基因为目标的检测策略,在保证检测精度的同时,大幅缩短检测时间并降低成本。
这些特定的对立等位基因称为“STR(短串联重复序列)”,在扩增后被用于比对,以确认亲属关系(如父子关系、亲子关系、兄弟关系等)。
什么是 STR?
STR(短串联重复序列;Short Tandem Repeats)是指在 DNA 序列中连续重复出现的、由相同短序列单元组成的片段。
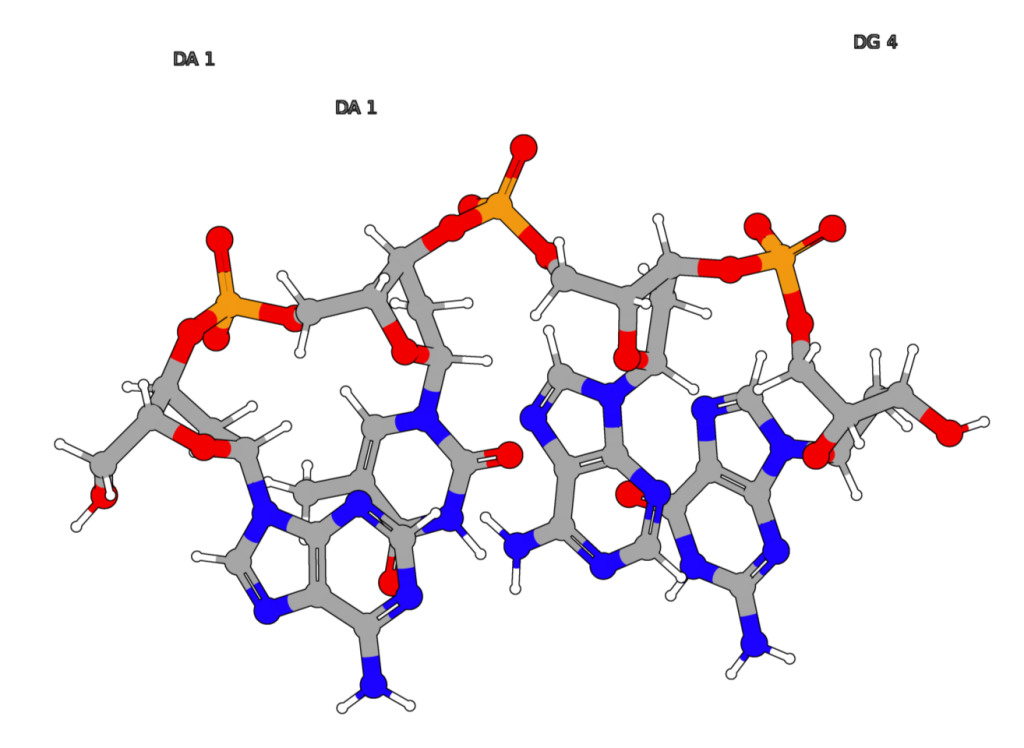
图1:以名为 CSF1PO 的 STR 为例,由 AGAT 这一短序列组成,并重复 5 到 17 次。
图2:D3S1358、D4S2408、FGA 是三个 STR 的示意图。虽然 STR 的种类很多,但每个人基本上在每种类型上都有两个,一个来自父亲,一个来自母亲。
STR的分析方法
具有特定多态性的短串联重复序列(STR)基因位点,会经过两轮聚合酶链式反应(PCR)进行标记,然后进行靶向型下一代测序(NGS)分析。该方法具备极高的读取深度和灵敏度,因此即便是使用过的牙刷、吸管等样本,也能够成功提取并检测DNA。
本检测不仅涵盖了美国联邦调查局(FBI)“联合DNA索引系统”(CODIS)核心的20个常染色体STR,还扩展至共27个常染色体STR位点,同时也包括了性别判定的标记基因“牙釉蛋白基因”(Amelogenin),以及25个Y染色体STR,从而实现更全面、更高精度的分析。
虽然牙釉蛋白基因在法医学与产前检测中已被广泛应用于性别判断,但本检测通过额外引入25个Y染色体标记,使性别判定的准确性和可信度进一步提高。
我们采用的是全球首个专为法医学基因组学设计、开发与验证的下一代测序平台——MiSeq FGx测序系统。在完成该系统中高精度遗传面板的测序后,将借助集成的Universal Analysis Software(UAS)进行数据分析。该平台采用专有的比对算法,对正向与反向引物序列及STR重复区域周边区域进行检视。它不是依据扩增子的长度来判断,而是利用整个扩增子序列,来准确识别各基因座、等位基因及个体基因型。
在分析过程中,低于特定阈值的读取数(read count)的基因位点将被排除,而高于阈值的读取会被作为等位基因进行判断。此外,系统还综合考虑群体中的等位基因频率、连锁不平衡、同一染色体上多个STR的分布等关键因素,对测序数据进行严格解读,从而确保检测结果的稳健性、灵敏度与准确性。
所有所用的系统与试剂盒均经过验证测试,符合欧洲标准、SWGDAM(科学工作组DNA分析方法指南)、CODIS(联合DNA索引系统)、国际刑警组织(Interpol)及欧洲标准系统(ESS)等最低规范要求。
至于通过UAS(Universal Analysis Software)执行的生物信息分析,以及用于亲属关系鉴定的统计学算法,属于本说明之外的内容。若您对数学细节感兴趣,可参考相关文献,以了解所用分析方法的概要。
- Kane K. (1982). Paternity exclusion and probability of paternity. Annals of clinical and laboratory science, 12(4), 309–314.
- Bugert, P., Rink, G., Kemp, K., & Klüter, H. (2012). Blood Group ABO Genotyping in Paternity Testing. Transfusion medicine and hemotherapy : offizielles Organ der Deutschen Gesellschaft fur Transfusionsmedizin und Immunhamatologie, 39(3), 182–186. https://doi.org/10.1159/000339235
- S. Arnott, P.J. Campbell-Smith & R. Chandrasekaran. Handbook of Biochemistry and Molecular Biology, 3rd ed. Nucleic Acids–Volume II, G.P. Fasman, Ed. Cleveland: CRC Press, (1976). pp. 411-422.
- Schrödinger, L., & DeLano, W. (2020). The PyMOL Molecular Graphics System, Version 3.1.0a0. Schrödinger, LLC.. Retrieved from http://www.pymol.org/pymol
- Tomasello, Gianluca, et al. ‘The Protein Imager: A Full-Featured Online Molecular Viewer Interface with Server-Side HQ-Rendering Capabilities’. Bioinformatics, edited by Arne Elofsson, vol. 36, no. 9, May 2020, pp. 2909–11. DOI.org (Crossref), https://doi.org/10.1093/bioinformatics/btaa009.
- Stephens, Kathryn M., et al. ‘Developmental Validation of the ForenSeq MainstAY Kit, MiSeq FGx Sequencing System and ForenSeq Universal Analysis Software’. Forensic Science International: Genetics, vol. 64, May 2023, p. 102851. DOI.org (Crossref), https://doi.org/10.1016/j.fsigen.2023.102851.
- Thangaraj, K., Reddy, A. G., & Singh, L. (2002). Is the amelogenin gene reliable for gender identification in forensic casework and prenatal diagnosis?. International journal of legal medicine, 116(2), 121–123. https://doi.org/10.1007/s00414-001-0262-y
- Jäger, AC, et al. Developmental validation of the MiSeq FGx Forensic Genomics System for targeted next generation sequencing in forensic DNA casework and database laboratories. Forensic Sci Int Genet. 2017; 28: 52–70. doi.org/10.1016/j.fsigen.2017.01.011.
- Nakazato T, Ohta T, Bono H. Experimental design-based functional mining and characterization of high-throughput sequencing data in the sequence read archive. PLoS One. 2013; 8 (10): e77910. doi.org/10.1371/journal.pone.0077910.
- Ballard D, Winkler-Galicki J, Wesoły J. Massive parallel sequencing in forensics: advantages, issues, technicalities, and prospects. Int J Legal Med. 2020; 134: 1291–1303.doi.org/10.1007/s00414-020-02294-0.
- Ingold S, et al. Body fluid identification using a targeted mRNA massively parallel sequencing approach – results of a EUROFORGEN/EDNAP collaborative exercise. Forensic Sci Int Genet. 2018; 34: 105–115. doi.org/10.1016/j.fsigen.2018.01.002.
- Bentley, DR, et al. Accurate whole human genome sequencing using reversible terminator chemistry. Nature. 2008; 456: 53–59. doi.org/10.1038/nature07517.
- Huston, K. A. (1998). Statistical analysis of STR data. Profiles DNA, 1(3), 14-15.
- Evett, Ian, and B. S. Weir. Interpreting DNA Evidence: Statistical Genetics for Forensic Scientists. Sinauer Associates, 1998.
- Gjertson, D. W., Brenner, C. H., Baur, M. P., Carracedo, A., Guidet, F., Luque, J. A., … Morling, N. (2007). ISFG: Recommendations on biostatistics in paternity testing. Forensic Science International: Genetics, 1(3-4), 223–231. doi:10.1016/j.fsigen.2007.06.006






