
この記事の概要
年齢を重ねると気になってくる「ひざの痛み」。実はそれ、生活習慣だけでなく「遺伝子」が関係しているかもしれません。最近の国際的な研究で、GDF5(成長分化因子5)という遺伝子のある変異が、膝関節症のリスクを高めることが統計的にも明らかになりました。本記事では、この遺伝子とひざの健康との関係を、わかりやすく解説します。
背景|Background
膝関節症(knee osteoarthritis)は、世界中の中高年層に多くみられる最も一般的な筋骨格系疾患の一つであり、関節軟骨の進行性の変性、骨の再構築、炎症反応を特徴とし、慢性的な疼痛やこわばり、可動域の制限、最終的には障害を引き起こします。この疾患は、医療機関の受診件数、人工関節置換術の施行件数、長期ケアの需要などを通じて、保健医療資源に大きな負担を与えています。

膝関節症の発症には、加齢変化、機械的負荷、炎症、代謝異常(例:肥満)などの複数の因子が関与しますが、近年では遺伝的素因の重要性が強調されています。双子研究や家族集積性の研究からは、膝関節症の感受性の40〜65%が遺伝的要因に起因すると示唆されています。これまでに数多くのゲノムワイド関連解析(GWAS: genome-wide association study)が行われ、疾患感受性に関与する特定の遺伝子や遺伝子多型の同定が進められてきました。
その中でも特に注目されているのが、GDF5(Growth Differentiation Factor 5)遺伝子であり、この遺伝子は関節や骨格系の発生・維持に不可欠なタンパク質をコードしています。本研究は、GDF5遺伝子内の特定の一塩基多型(SNP: single nucleotide polymorphism)であるrs143383が、膝関節症のリスクと関連しており、その関連がゲノムワイド有意水準(p < 5×10⁻⁸)を超えるかどうかを検証することを目的としています。
関連遺伝子&SNP(一塩基多型)|Associated genes & SNPs
GDF5(成長分化因子5)遺伝子は、TGF-β(Transforming Growth Factor-beta)スーパーファミリーに属するシグナル伝達分子の一つであり、細胞の増殖・分化・組織形成の調節に関与します。GDF5は特に骨端成長板、関節軟骨、滑膜構造など、骨や関節の発生・維持に重要な部位で高発現しています。動物モデルでは、GDF5が靱帯、腱、骨の損傷治癒を促進することも示されています。

rs143383は、GDF5遺伝子の5’非翻訳領域(5′ UTR)に位置する機能的SNPです。5′ UTRは翻訳を開始する前の遺伝子配列であり、mRNAの安定性や翻訳効率を調節する役割を担っています。この多型はT(チミン)とC(シトシン)の置換によって構成され、Tアレルを持つ個体ではGDF5の発現量が有意に低下することが分かっています。その結果、関節構造の維持に必要なGDF5タンパク質の量が減少し、関節症のリスクが上昇することが考えられます。
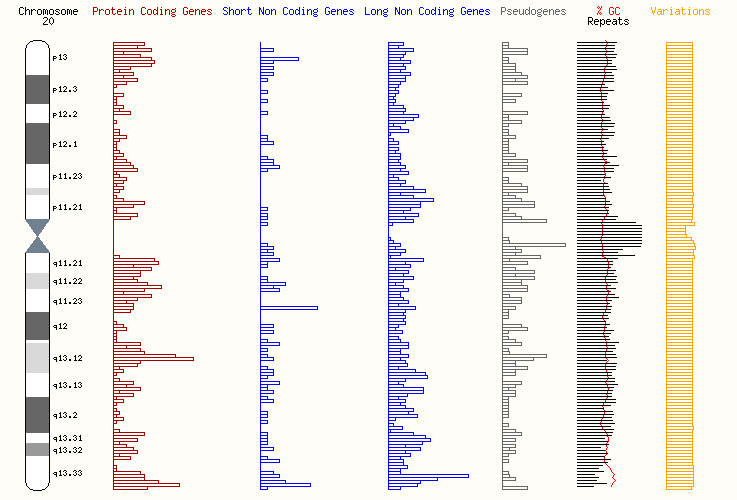
このrs143383 Tアレルは、これまでアジア人集団での膝関節症との関連が複数報告されており、再現性の高い遺伝的リスク因子の一つとされています。ただし、欧州系集団ではその効果が比較的小さい傾向にあるため、多民族集団を対象とした大規模メタ解析が求められていました。
考察:この研究から何が分かったのか?|Discussion
本研究は、GDF5遺伝子のプロモーター領域に存在するrs143383多型が、膝関節症のリスクと有意に関連することを明らかにしました。特に、Tアレルを持つ個体では膝関節症の発症リスクが17%増加することが示され(オッズ比(OR)1.17、95%信頼区間(CI)1.12〜1.23)、この関連は統計学的にゲノムワイド有意水準(p = 6.2×10⁻¹¹)を満たしています。さらに、解析対象を欧州系集団に限定しても同様の関連が確認され(p = 8.3×10⁻⁹)、人種に依存しない普遍的なリスク因子である可能性が示唆されました。

感度分析として、ハーディ・ワインベルグ平衡(Hardy–Weinberg equilibrium)に違反していた1研究を除外した場合、関連性はさらに強まり(OR 1.18、p = 4.1×10⁻¹¹)、本結果の頑健性が裏付けられました。
また、性別による層別解析では、女性における効果量(OR 1.19)が男性(OR 1.14)よりやや大きい傾向がみられましたが、有意差は認められず、性差を問わず安定した関連性が示されました。さらに、研究間の異質性(heterogeneity)は検出されず、結果の一貫性と信頼性が強調されました。
以上から、GDF5 rs143383は機能的な遺伝的多型であり、膝関節症の遺伝的素因として強力かつ再現性の高い証拠を持つことが明確になりました。今後のリスク予測や治療標的の検討において、GDF5の役割は重要なものとなるでしょう。
研究方法|Methods
本研究では、英国、エストニア、オランダの3か国から得られた複数の独立コホートを統合し、膝関節症とrs143383の関連を検討しました。具体的には、新たに収集された膝関節症患者2,235例および対照群3,303例のデータを用い、さらにChingford Study(259例の患者、509例の対照)および過去に発表された大規模メタ解析の結果を加え、全体の解析対象を拡大しました。

英国およびエストニアのサンプルは、Kbioscience社(英国ヘリフォードシャー)により既存の手法で遺伝子型解析が実施されました。ロッテルダム研究(Rotterdam Study III)では、すでに構築されたゲノムワイド関連データベースから該当データが抽出されました。放射線診断や臨床評価の手法については、先行文献で詳細に報告されています。
最終的に、欧州系被験者からは10,103名の対照と6,861名の膝関節症患者、アジア系被験者からは1,844名の対照と718名の患者を含む、十分な統計学的検出力(86%)を備えたデータセットが構築されました(効果量OR 1.15以上を検出可能)。

すべての研究では、倫理審査委員会による承認が取得され、参加者からのインフォームド・コンセントが得られています。
研究結果|Results
メタ解析の結果、rs143383 Tアレルが膝関節症のリスク増加と有意に関連することが示されました。全サンプルでのオッズ比は1.17(95%CI 1.12〜1.23)、p値は6.2×10⁻¹¹に達しました。欧州系被験者のみでの解析においてもp = 8.3×10⁻⁹と、ゲノムワイド有意水準(p < 5×10⁻⁸)を大きく下回りました。

Hardy–Weinberg平衡に違反していた1研究を除外した場合、関連性はさらに強まり(OR 1.18、p = 4.1×10⁻¹¹)、関連の確実性が増しました。性別ごとの分析では、男性でのORが1.14(p = 1.6×10⁻³)、女性でのORが1.19(p = 3.7×10⁻⁶)と、両群において有意な関連が認められました。

各研究におけるアレル頻度やORも報告されており、例えばChingford Studyでは、Tアレル頻度が患者群で67.6%、対照群で59.5%、オッズ比は1.42(95% CI 1.13〜1.77)と示されています。これらの結果は、研究間での効果方向が一貫していることを支持しています。
結論|Conclusion
本研究により、GDF5遺伝子のrs143383多型が膝関節症のリスク因子として信頼性の高い関連を持つことが、ゲノムワイド有意水準を満たす形で明らかになりました。この関連は複数の独立したコホートで再現され、性別や人種に関わらず一貫して観察されました。今後の遺伝的リスク評価、個別化医療、ならびにGDF5経路を標的とした治療戦略の開発において、本多型は重要なマーカーとなる可能性があります。

キーワード|Keywords
GDF5, rs143383, 膝関節症, ゲノムワイド関連解析, 遺伝的リスク因子, 一塩基多型
GDF5, rs143383, knee osteoarthritis, genome-wide association study, genetic risk factor, single nucleotide polymorphism (SNP)

引用文献|References
- Valdes, A. M., Evangelou, E., Kerkhof, H. J., Tamm, A., Doherty, S. A., Kisand, K., Tamm, A., Kerna, I., Uitterlinden, A., Hofman, A., Rivadeneira, F., Cooper, C., Dennison, E. M., Zhang, W., Muir, K. R., Ioannidis, J. P., Wheeler, M., Maciewicz, R. A., van Meurs, J. B., Arden, N. K., … Doherty, M. (2011). The GDF5 rs143383 polymorphism is associated with osteoarthritis of the knee with genome-wide statistical significance. Annals of the rheumatic diseases, 70(5), 873–875. https://doi.org/10.1136/ard.2010.134155
- The UniProt Consortium, Bateman, A., Martin, M.-J., Orchard, S., Magrane, M., Adesina, A., Ahmad, S., Bowler-Barnett, E. H., Bye-A-Jee, H., Carpentier, D., Denny, P., Fan, J., Garmiri, P., Gonzales, L. J. D. C., Hussein, A., Ignatchenko, A., Insana, G., Ishtiaq, R., Joshi, V., … Zhang, J. (2025). Uniprot: The universal protein knowledgebase in 2025. Nucleic Acids Research, 53(D1), D609–D617. https://doi.org/10.1093/nar/gkae1010
- Jumper, J., Evans, R., Pritzel, A., Green, T., Figurnov, M., Ronneberger, O., Tunyasuvunakool, K., Bates, R., Žídek, A., Potapenko, A., Bridgland, A., Meyer, C., Kohl, S. A. A., Ballard, A. J., Cowie, A., Romera-Paredes, B., Nikolov, S., Jain, R., Adler, J., … Hassabis, D. (2021). Highly accurate protein structure prediction with AlphaFold. Nature, 596(7873), 583–589. https://doi.org/10.1038/s41586-021-03819-2
- Varadi, M., Bertoni, D., Magana, P., Paramval, U., Pidruchna, I., Radhakrishnan, M., Tsenkov, M., Nair, S., Mirdita, M., Yeo, J., Kovalevskiy, O., Tunyasuvunakool, K., Laydon, A., Žídek, A., Tomlinson, H., Hariharan, D., Abrahamson, J., Green, T., Jumper, J., … Velankar, S. (2024). AlphaFold Protein Structure Database in 2024: Providing structure coverage for over 214 million protein sequences. Nucleic Acids Research, 52(D1), D368–D375. https://doi.org/10.1093/nar/gkad1011
- Cheng, J., Novati, G., Pan, J., Bycroft, C., Žemgulytė, A., Applebaum, T., Pritzel, A., Wong, L. H., Zielinski, M., Sargeant, T., Schneider, R. G., Senior, A. W., Jumper, J., Hassabis, D., Kohli, P., & Avsec, Ž. (2023). Accurate proteome-wide missense variant effect prediction with AlphaMissense. Science, 381(6664), eadg7492. https://doi.org/10.1126/science.adg7492
- Perez, G., Barber, G. P., Benet-Pages, A., Casper, J., Clawson, H., Diekhans, M., Fischer, C., Gonzalez, J. N., Hinrichs, A. S., Lee, C. M., Nassar, L. R., Raney, B. J., Speir, M. L., van Baren, M. J., Vaske, C. J., Haussler, D., Kent, W. J., & Haeussler, M. (2025). The UCSC Genome Browser database: 2025 update. Nucleic acids research, 53(D1), D1243–D1249. https://doi.org/10.1093/nar/gkae974
- Harrison, P. W., Amode, M. R., Austine-Orimoloye, O., Azov, A. G., Barba, M., Barnes, I., Becker, A., Bennett, R., Berry, A., Bhai, J., Bhurji, S. K., Boddu, S., Branco Lins, P. R., Brooks, L., Ramaraju, S. B., Campbell, L. I., Martinez, M. C., Charkhchi, M., Chougule, K., … Yates, A. D. (2024). Ensembl 2024. Nucleic Acids Research, 52(D1), D891–D899. https://doi.org/10.1093/nar/gkad1049


