使用阵列比较基因组混合翻译猫叫综合症中基因型-表型关系的高分辨率映射论文 (Hiro诊所NIPT初步翻译)
1 加利福尼亚大学旧金山分校综合癌症中心·实验室医学系、2 加利福尼亚大学旧金山分校癌症研究所、3 奥胡斯大学人类遗传学研究所 (丹麦奥胡斯)、4 哥本哈根大学医学生物化学和遗传学研究所 (哥本哈根)、5 北京市中国科学院北京基因组研究所
简介
染色体5p的缺失导致各种发育缺陷,大多数病例被归类为猫叫综合症 (MIM123450) (Niebuhr 1978a) 。这些缺失是末端的或间质的,有时与细胞遗传学上复杂的核型有关(Sreekantaiah et al.1999).猫叫综合症具有几种表型元件,例如特征性的哭声,面部异常,声音延迟,精神发育迟滞 (MR) ,从而给出该综合症的名称。
一些先前的研究显示5p上缺失片段的范围与表型之间的关系 (Overhauser等1994;Church等1997;Marinescu等1999a;Mainardi等2001) 。然而,这些研究导致关于MR与已删除区域的关系的一些不一致的结果和存在物质争议。不同的结果很可能是由于多种因素的组合造成的,这些因素包括多个观察者对表型的不一致评估、缺乏对某些表型特征的显性的年龄依赖性的考虑、以及用于评估基因型的分析技术的限制(Wilkinset al.1983;Church et al.1995;Marinescu et al.1999 a;Johnson et al.2000)。在这项研究中,所有这些潜在问题都被涵盖。
在先前的基因型分析中,已经使用常规细胞遗传学、FISH、多态性标记和最近基于染色体的比较基因组杂交 (CGH) 。(Marinescu et al.1999 b;Levy et al.2002)检测并定义异常的性质。
传统的细胞遗传学分析提供了全基因组的概述,但分辨率有限。因此,像差的边界设置不正确,小的缺失被忽略,复杂的异常被不适当地识别。尽管FISH和多态性标记物允许对基因组序列的异常的精确评估和异常的映射,但是由于探针必须单独或以少数组进行评估,因此需要大量的精力。因此,对多个基因座和样品的研究是非常费力的。染色体CGH可以在单个杂交中筛选DNA拷贝数变化的整个基因组,但其分辨率限制为约5~10 Mb,并且结果不能直接映射到基因组序列上。
最近,笔者开发了一种实施微阵列CGH的方法 (Solinas-Toldoet al.1997;Pinkel et al.1998;Pollack et al.1999;Snijders et al.2001;Fiegler et al.2003) 。本阵列使用由大插入基因组复制 (例如BACS或PACS) 产生的阵列元件,并且具有足够的测量精度以允许对影响单个克隆体的单个复制异常的可靠检测。使用诸如STS或末端序列的序列标签来确定基因组序列上每个克隆体的位置。通过这种阵列获得的分辨率由克隆体之间的基因组间隔和克隆体长度确定。通过使用重复复制 (Albertson et al.2000) ,可以获得BAC长度的一些分辨率。
在该研究中,使用常规细胞遗传学和FISH将阵列CGH应用于分析确定具有5p缺失的94名患者的基因组DNA。在我们进行分析之前,从接受了符合标准的详细表型评估的5p缺失病例的积累中选择这些样本 (Niebuhr 1978 b);笔者等人未发表的结果) 。大多数患者都在几岁时进行评估,以便可以在评估最好的发展阶段建立表型。这些病例的子集已包含在5p缺失的影响和基因型-表型相关性的先前研究中(Overhauser et al.1990, 1994;Church et al.1995、1997).本研究的结果显示了阵列CGH用于评估这些患者的重要性,使得能够揭示和改善猫叫综合症中MR,语音延迟,面部异形和特征性哭声的基因型-表型相关性。
方法
猫叫综合症患者
受试者选自150多名5p缺失患者的组,这些患者在过去几十年中由其中1人 (E.N.) 进行了广泛研究。如果可以获得详细的临床数据和基因组DNA,将受试者纳入其中。为了便于进一步的研究,作者提供了关于体细胞与异常染色体和异常5号染色体的杂合体的亲源的先前研究,并且仅分析了可获得家族基因组DNA的受试者。对于大多数受试者,常规细胞遗传学分析的结果是可用的。表1列出了94个合格的受试者,包括表型评估、常规细胞遗传学评估和阵列结果。患者编号1~69是指丹麦的持续 (诊断时) 家庭成员。 超过100名患者分别来自其他国家,包括来自挪威的100-117名患者、来自英国的201-256名患者、来自捷克斯洛伐克的300名患者和来自澳大利亚的402名患者,并且已经被介绍用于更详细的临床和细胞遗传学评估。因此,这些病例是具有5p缺失的个体的准随机取样。这些样本的阵列CGH测量是在获得加州大学旧金山分校的试验审查委员会的适当批准后进行的。
通过单个观察者(E.N.)评估表型特征,并统一应用分类标准。由于表型方面可能在发育期间发生变化,因此大多数患者在5岁以下或5岁以上时重新评估。表型特征的存在与否在表1中分别用「Y」 或 「N」 标记。因为数据的缺失显示为「ND」 ,且表型特征可能取决于年龄,所以如果任何年龄组的受试者都存在 「Y」 ,则将表型特征归类为阳性。例如,哭声和面部形态异常在患者年轻时是最明显的,因此在老年组中这些表型的丧失并不被认为是重要的。 某些患者在某一年龄范围内没有表现出特定的表型 (表示「N」 ) ,而其他患者无法获得数据,导致表型状态的不确定性。这些病例被排除在确定其特征和基因型之间的关系之外。因此,患者18、45、56、112和117不用于定义表皮区域,并且患者56和112也不用于确定与面部畸形相关的基因组区域。具有最严重精神障碍的患者没有用于确定言语延迟区域,因为可能由于其他原因而导致言语能力受损。因此,没有使用患者45来定位表型声音分量。
定义MR严重程度的精神活动发展评估是基于个人观察 (E.N.) ,文件信息,心理测试,学校成绩信息等。由于MR在幼儿中难以评估,因此仅使用5岁以上数据与缺失相关的患者。因此,将22例、225例和231例排除在本分析之外。MR的程度由0 (不受影响) 到7 (严重影响) 范围的数值标度表示。各分类的一般判定标准如表2所示。这些评估的不确定性估计在±0.5和±1.0之间的范围内,在量表的底部和顶部。
用于阵列-5 p的高分辨率分析的阵列包括由使用遗传映射的STS标记选择的BAC、P 1和PAC克隆产生的元件(Hudson et al.1995;Peterson et al.1999),以及在用于正常化和其它异常检测的其它染色体上的可变数量的复制。所有复制的位置都基于2003年7月加利福尼亚大学圣克鲁兹 (UCSC) 冻结的人类基因组。5p的覆盖密度在先前定义为猫叫综合症的重要区域中最高。表3显示了55个克隆体,包括在这些患者中观察到的所有5p异常。当基因组序列完成后,我们可以准确地绘制大多数克隆体。
然而,一些序列位置必须根据遗传地图信息进行插补。
在阵列上分析所有患者,包括5p上的55个克隆体和其他位置上的100~300个克隆体。对于各种版本的阵列,包括分布在整个基因组中的约1,750~2,000个克隆体,对其中37名患者进行分析 (Snijders et al.2001) 以检查涉及其他染色体的异常。其中一些较大的阵列不包含一套完整的5p克隆体。
从克隆体中分离出阵列元件的DNA,并通过连接体适配器PCR进行扩增。使用定制打印机将PCR产物点染到涂有铬的载玻片上,该定制打印机使用毛细管印刷销 (该作者未发表的材料) 悬浮在浓度为约0.8μg/μl的水中20%二甲基亚砜上。为每个克隆体打印了三个相邻的斑点。对于较小的阵列,在阵列上广泛分离的位置打印两组三联。
阵列CGH杂交和分析-使用标准技术从患者和对照受试者的外周血中分离全基因组DNA,并使用任何缺口翻译 (1μg) 标记DNA (Pinkel et al.1998)或随机引物延伸 (0.5μg) (Snijders et al.2001)。标记使用与dCTP直接结合的荧光染料。一些测量使用荧光素和德克萨斯红 (或Alexa 594) ,而其它测量使用Cy3和Cy5分别标记测试基因组和参考基因组。
用含有50μg的Cot -1 DNA来抑制重复序列混合的标记测试和参考基因组DNA被乙醇沉淀并再悬浮于杂化混合物中,以形成50%甲酰胺/10%硫酸葡聚糖/2#SSC/1%-4% (v/v) SDS的最终组成,总体积为50μl。
将杂化混合物加热至70°C以使DNA变性,并将温度降低至37°C约1小时以允许重复序列的再聚集。为了混合,在阵列周围形成了由橡胶水泥制成的低屏障。然后,将杂交混合物施加到阵列上,并将阵列置于密封室中,其中流体的上表面保持自由。混合在37°C缓慢摇动的工作台上进行16至48小时 (Pinkel et al.1998) 。
混合后,将阵列置于PN缓冲液 (0.1M磷酸钠) 中,以除去大部分混合溶液;0.1%诺尼迪特P 40;在pH 8) 的流动中快速洗涤,然后在45°C下在50%甲酰胺/2#SSC中浸渍15分钟,然后在室温PN缓冲液中最终洗涤15分钟。清洗阵列后,将含有1μM DNA染色DAPI (4′,6-二氨基-2-苯基吲哚)的90%甘油和10%磷酸缓冲液pH 9施用于每个阵列,并加入盖玻片。DAPI对阵列斑点进行染色并使其可视化,而不考虑混合信号。
阵列用定制电荷耦合器件 (CCD) 成像系统(Pinkel et al.1998;本文作者未发表的数据) 成像。整个阵列包含在一个CCD图像中。大多数分析使用图像分析软件UCSF SPOT (Jain et al.2002)。该程序使用DAPI图像来确定阵列中的点的位置,并在减去局部背景之后计算每个点中的每个像素的测试和参考强度。使用总测试强度 (减去背景) 与总参考强度 (减去背景) 之比作为每个点的相对复制数的指标,并且平均复制比率。不对图像或比率数据进行任何类型的计算调整(阴影校正、低规格化、空间依赖性规格化和克隆特异性规格化) 。为了识别问题信号,应用了质量标准,包括点中的测试信号和参考信号之间的相关性。通常,从分析中去除具有相关r<0.8个地点被拒绝了。关于副本的SD>0.2的克隆。在大多数情况下,复制点的SD<0.02。我们还放弃了对克隆的测量,其中只有一个克隆经过质量检验。应用整体归一化系数,将每个单元的两个副本的阵列元素的log2比率的中值 (即中值线性比率) 分别设置为0或1。
图1
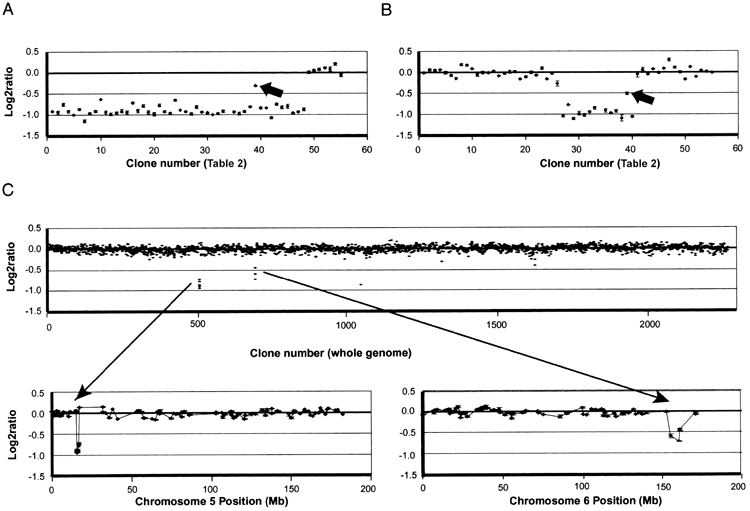
表1 94例猫叫综合症患者主要数据摘要
| 患者 | 表型 | 级别 | 传统细胞遗传学a | 边界 (Mb/clone no.b) |
|||
|---|---|---|---|---|---|---|---|
| 特征性的哭声c | 面部畸形c | 声音延迟 | 近端d | 远端 | |||
| 1 | Y/Y | Y/Y | Y | 5 | 46,XY,del(5)(p14.3)* | 20.837/39 | |
| 3 | Y/Y | Y/Y | Y | 6.5 | 46,XY,del(5)(p13.3)* | 30.726/51 | |
| 4 | Y/Y | Y/Y | Y | 6 | 46,XY,der(5)t(5;22)(p14.2;p13)pat | 30.022/44 | |
| 5 | Y/Y | Y/Y | Y | 5.5 | 46,XY,del(5)(p14.3)* | 17.500/37 | |
| 6 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 23.276/41 | |
| 8 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p14.1)* | 25.841/42 | |
| 9 | Y/Y | Y/Y | Y | 7 | 45,XX,dic(5;13)(p13.3;p12)* | 31.942/45 | |
| 10 | Y/Y | Y/Y | Y | 5.5 | 46,XX,del(5)(p14.2)* | 21.797/40 | |
| 11 | ND/Y | Y/Y | Y | 5.5 | 46,XX,del(5)(p14.1)* | 30.022/44 | |
| 12 | ND/Y | Y/Y | Y | 5.5 | 46,XX,del(5)(p14.3)* | 18.336/38 | |
| 13 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p13.3)* | 33.428/49 | |
| 14 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p13.3)* | 30.022/44 | |
| 15 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.3)* | 20.837/39 | |
| 16 | Y/Y | Y/N | Y | 6.5 | 46,XX,der(5)t(5;8)(p14.1;p22)* | 31.751/46 | |
| 17 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 25.841/42 | |
| 18 | ND/N | Y/N | N | 6 | 46,XY,del(5)(p14.1p15.31)*IN | 30.022/44 | 8.021/15 |
| 20 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 31.751/46 | |
| 21 | Y/Y | Y/Y | Y | 5.5 | 46,XX,del(5)(p14.3)* | 18.336/38 | |
| 22 | Y/ND | Y/ND | Y | NAe | 46,XX,add(5)(p14.3)* | 21.797/40 | |
| 23 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p14.1)* | 30.022/44 | |
| 24 | Y/Y | Y/N | Y | 7 | 46,XX,add(5)(p14.1)mat | 21.797/40 | |
| 25 | Y/Y | Y/N | Y | 7 | 46,XX,add(5)(p14.2)* | 18.336/38 | |
| 26 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p14.1)* | 31.751/46 | |
| 27 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p13.3)* | 33.428/49 | |
| 28 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.3)* | 20.837/39 | |
| 30 | Y/Y | Y/Y | Y | 5.5 | 46,XX,del(5)(p14.3)* | 20.837/39 | |
| 31 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p13.3)* | 31.942/45 | |
| 35 | Y/ND | Y/ND | Y | 6.5 | 45,XY,der(5)t(5;14)(p14.2;q12),-14,mat | 23.276/41 | |
| 36 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 30.022/44 | |
| 37 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 30.022/44 | |
| 38 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p15.1)* | 18.336/38 | |
| 39 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 30.022/44 | |
| 40 | Y/Y | Y/Y | Y | 5 | 46,XY,del(5)(p15.1)* | 16.684/33 | |
| 42 | Y/Y | Y/ND | Y | 3 | 46,XX,del(5)(p15.2)* | 11.035/28 | |
| 44 | Y/Y | Y/ND | Y | 3 | 46,XX,del(5)(p15.2)* | 11.035/28 | 1.207/1 |
| 45 | ND/N | N/N | Y | 7 | 46,XY,der(14)t(5;14)(q11;p13) del(5)(p14.3p15.31)* | 17.500/37 | 14.929/30 |
| 48 | Y/N | Y/N | Y | 6.5 | 46,XY,add(5)(p14.2)* | 16.684/33 | |
| 49 | Y/Y | Y/Y | N | 5 | 46,XX,der(5) ins(15;5)(q22;p14.3p15.31)mat | 26.794/43 | 5.945/12 |
| 50 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 31.942/45 | |
| 51 | N/N | N/N | N | 0 | 46,XX,del(5)(p13.3p14.1)mat | 31.942/45 | 18.336/38 |
| 52 | Y/Y | Y/Y | Y | 6.5 | 46,XY,del(5)(p13.3)* | 31.751/46 | |
| 54 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p15.1)* | 16.684/33 | |
| 55 | Y/Y | Y/Y | Y | 5.5 | 46,XX,der(5)t(5;13)(p14.3;p12)mat | 20.837/39 | |
| 56 | ND/N | ND/N | ND | 6 | 46,XY,der(5)t(5;7)(p14.1p21)? del(5)(p14.1p15.1)?del(7)* | 26.794/43 | 8.021/15 |
| 58 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.1)* | 30.022/44 | |
| 59 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.3)* | 23.276/41 | |
| 60 | Y/Y | Y/Y | Y | 5 | 46,XY,del(5)(p14.2)* | 20.837/39 | |
| 63 | Y/Y | Y/Y | Y | 5 | 46,XX,rec(5)del(5)(p14.2) inv(5)(p14.1p15.1)mat | 21.797/40 | |
| 66 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p14.2)* | 23.276/41 | |
| 68 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p14.1)* | 25.841/42 | |
| 101 | Y/Y | Y/ND | Y | 2.5 | 46,XX,del(5)(p15.2)* | 11.035/28 | |
| 102 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.2p15.33)* | 23.276/41 | 3.174/4 |
| 104 | Y/Y | Y/Y | Y | 6.5 | 46,XY,del(5)(p13.3)* | 32.162/48 | |
| 105 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.3)* | 17.224/36 | |
| 107 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p13.3)* | 33.428/49 | |
| 109 | Y/Y | Y/Y | Y | 5.5 | mos 46,XX,del(5)(p14.1)/del(5)(p15.2)* | 25.841/42 | |
| 110 | Y/Y | Y/Y | Y | 6.5 | 46,XX,del(5)(p13.3)* | 30.726/51 | |
| 111 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p14.1)* | 25.841/42 | |
| 112 | ND/N | ND/N | N | 1 | 46,XX,del(5)(p14.2p15.2)* | 23.276/41 | 11.435/27 |
| 113 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.1)* | 23.276/41 | |
| 114 | N/N | N/N | N | 2 | 46,XY,del(5)(p14.2p15.2)* | 23.276/41 | 11.360/26 |
| 115 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p15.2)* | 11.035/28 | |
| 117 | ND/N | N/N | Y | 3.5 | 46,XY,del(5)(p15.31)* | 9.288/18 | |
| 201 | Y/Y | Y/N | Y | 7 | 46,XY,add(5)(p14.3)* | 17.224/36 | |
| 202 | Y/Y | Y/ND | Y | 2.5 | 46,XY,del(5)(p15.2)* | 11.435/27 | |
| 203 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p13.3)* | 30.022/44 | |
| 205 | Y/Y | Y/Y | Y | 5 | 46,XY,del(5)(p14.2)* | 21.797/40 | |
| 206 | Y/Y | Y/ND | Y | 7 | 46,XX,add(5)(p14.3)pat | 18.336/38 | |
| 209 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p13.3)* | 31.751/46 | |
| 210 | Y/Y | Y/Y | Y | 7 | 46,XX,del(5)(p14.1)pat | 25.841/42 | |
| 212 | Y/Y | Y/Y | Y | 6.5 | 46,XY,del(5)(p14.1)* | 30.022/44 | |
| 213 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.2)* | 20.837/39 | |
| 214 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.2)* | 17.224/36 | |
| 215 | Y/Y | Y/N | Y | 7 | 46,XX,der(5)t(4;5)(q32;p14.3)* | 14.929/30 | |
| 216 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.2)* | 20.837/39 | |
| 218 | Y/Y | Y/Y | Y | 6.5 | 46,XX,add(5)(p14.3)pat | 17.500/37f | |
| 219 | Y/Y | Y/Y | Y | 6.5 | 46,XY,del(5)(p14.1)* | 25.841/42 | 1.207/1 |
| 221 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.2)* | 18.336/38 | |
| 222 | Y/Y | Y/Y | Y | 6 | 46,XY,del(5)(p14.1)* | 26.794/43 | |
| 223 | Y/Y | Y/Y | Y | 6 | 46,XX,del(5)(p13.3)* | 33.428/49 | |
| 225 | Y/ND | Y/ND | Y | 4g | 46,XY,del(5)(p14.3p15.31)* | 21.797/40 3.174/4 | |
| 228 | Y/Y | Y/Y | Y | 6 | 46,XY,add(5)(p15.2)pat | 11.035/28 | |
| 229 | Y/Y | Y/Y | Y | 7 | 45,XX,dic(5;22)(p13.2 p12)* | 33.428/49 | |
| 231 | Y/ND | Y/ND | Y | 3.5g | 46,XX,del(5)(p15.1)* | 15.679/31 | |
| 232 | Y/Y | Y/Y | Y | 6.5 | 46,XX,del(5)(p13.3)* | 31.942/45 | |
| 250 | Y/Y | Y/Y | Y | 5 | 46,XX,del(5)(p14.1)* | 26.794/43 | |
| 251 | Y/Y | Y/Y | Y | 6.5 | 46,XX,del(5)(p13.3)* | 30.022/44 | |
| 252 | N/N | N/N | N | 5 | 46,XY,del(5)(p15.1p15.31)* | 15.872/32 | 6.365/13 |
| 253 | Y/Y | Y/Y | Y | 5 | 46,XY,r(5)(14.3q35.3)* | 20.837/39 | |
| 254 | Y/Y | Y/Y | Y | 5 | 46,XY,del(5)(p14.3)* | 20.837/39 | |
| 255 | Y/Y | Y/Y | Y | 6.5 | 46,XY,add(5)(p14.2)* | 20.837/39 | |
| 256 | Y/Y | Y/Y | Y | 5.5 | 46,XX,del(5)(p13.3)* | 30.022/44 | |
| 300 | Y/Y | Y/Y | Y | 5 | 46,XY,del(5)(p14.1)* | 25.841/42 | |
| 402 | Y/Y | Y/ND | Y | 3 | 46,XY,del(5)(p15.2)* | 11.035/28 | |
结果
阵列CGH测量
5号染色体和全基因组扫描的代表性复制数曲线如图1所示。单一副本的缺失和均匀样本中的增益理想地分别具有-1.0和+0.58的log2比率。
在实践中,缺失的比率在-1~-0.7之间,这可能是由于来自高度重复序列信号的不完全抑制、交叉混合以及背景水平的不完全定义而偏离理想。通过使用设置为等于-0.4的log 2比率的简单阈值,与复制数的比率和干扰等级(如图1所示)之间的这种近似定量关系,可以非常可靠地检测缺失。在大多数情况下,在单一混合中实现了足以建立两个阵列克隆体之间的一个或两个间隔的缺失边界的精度。如果第一次测量干扰太大,无法识别与缺失边界相邻的克隆体,则重复测量。
由于没有提供比再混合更多的信息,因此没有进行色素反转法测定。有时,已经发现样本DNA的重新纯化改善了数据的质量,并且表明一些样本中的未知污染物影响一致标记或杂交基因组DNA的能力。
表1总结了94例患者的缺失数据、常规细胞遗传学分析和表型特征。患者使用先前编号 (Niebuhr 1978 a;与作者未发表的资料) 相对应,按编号顺序列出。
在82名受试者中,缺失是末端的,而在12名受试者中,缺失是间质性的,在5p处共有106个缺失边界。如下所详述,在受试者中的15例中,除了5p缺失之外,还观察到拷贝数异常。这些异常中的3个是与缺失边界相邻的5p序列的重复,但其他异常如表4所示包含5p以外的基因组区域。
根据作者的测量,发现了两个有趣的DNA序列特征的迹象。首先,检测到一个重要的断点集群。在这些患者中观察到的所有106个缺失边界都发生在染色体的远端约33Mb内,从而导致每兆碱基约3个边界的平均密度。
在94例中,有9例在5p 15.2的克隆体26、27和28的约0.5Mb区中具有远端或近端边界,每兆碱基产生约18个边界。
此外,如果该区域的克隆如2003年7月的阵列冻结所示进行排序,则缺失边界看起来很复杂,并且与边界相邻的克隆的比率存在振荡。在所有情况下,对克隆进行排序简化了缺失边界的表观结构。在 表3中,以修订的顺序列举克隆,根据基因组序列的集合表示碱基序列的位置。这些结果表明,该区域中可能存在一些序列基序,其促进异常形成并使基因组序列的适当集合复杂化。或者,如果序列组合是正确的,则该区域的缺失边界的簇具有反映局部序列特征的重复复数复制结构。较高密度的克隆覆盖率将可能减小该簇区域的大小,并且可能揭示5p处的其它断点簇。
我们发现的第二个序列特征是正常人基因组中5p上的序列重叠区。58个示例(例如,图1A和1B)中的数据表明,当克隆39明显缺失时,在正常区域中介于预期和缺失之间的log 2比率至
显示有-0.4。然而,当缺失向近端延伸以包括克隆体51和52的区域时,克隆体39上的比率降低到期望值。这表明克隆体39序列的相当一部分是在克隆体51和52之间以5p复制的,并且仅包括克隆体39的缺失代表从4个副本到3个副本 (log2 [3/4] 到-0.4) 的变化。
使用该BAC作为探针的FISH分析证实了这一假设,并且在5p中发现了两个接近的信号。人类基因组序列的一部分的冻结 (2002年6月,2002年11月,2003年4月) 显示了这种重叠的存在,并且显示了BAC中的标记位于20.791Mb和34.070Mb (2003年4月) 。
2003年7月的冻结并不意味着重复。这种类型的大量重叠在基因组中频繁发生(Lupski et al.1996;Eichler 2001)
在该组中发现含有克隆体39的样品,其中DNA拷贝数没有多态性 (Albertson and Pinkel 2003;Iafrate et al.2004;Sebat et al.2004) 。该区域中可能的序列复杂性的进一步暗示是,如表3所述,序列集合与克隆顺序不一致,这是由关于克隆体51的缺失边界的最简单解释所指示的。
MR对缺失的依赖性
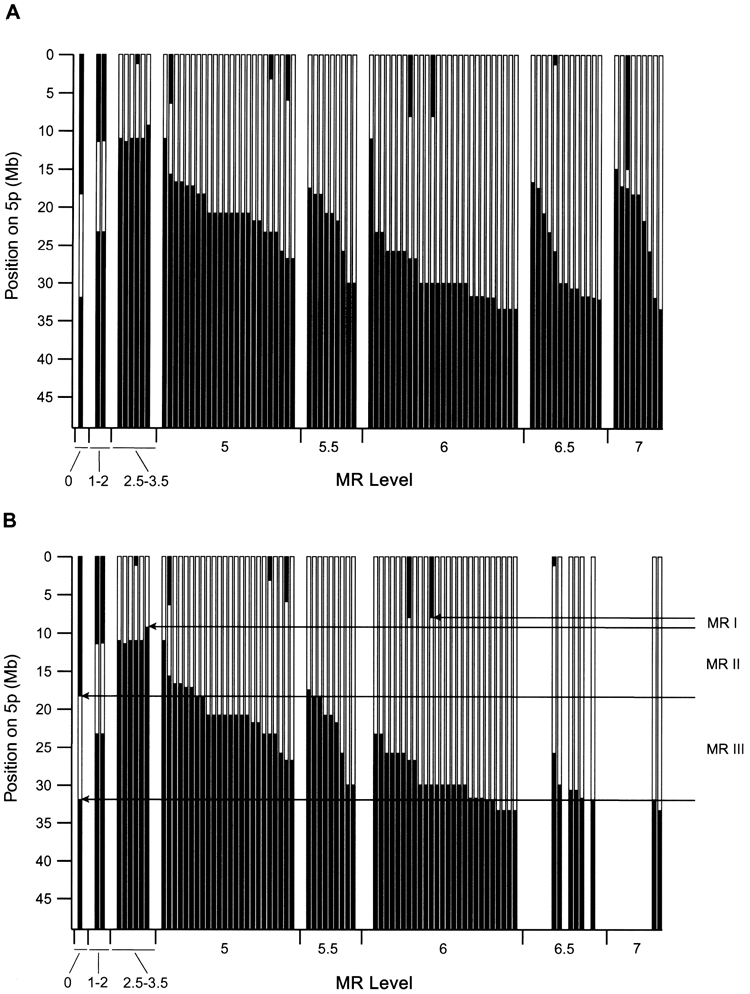
图2A示出了5p缺失与患者MR状态之间的关系,其中通过增加延迟水平对患者进行排序。这种关系看起来既复杂又有些尴尬。随着缺失程度的增加,延迟的严重程度明显增加,但也有一些重要的例外。例如,一些在5p处具有间质性缺失的患者不受影响或稍微延迟,而一些在相同区域具有小缺失的患者明显延迟。
先前已经对这些受试者进行了常规的细胞遗传学和FISH分析,并且已显示一些患者的染色体异常是复杂的以及除了5号染色体之外还涉及染色体重排。因此,使用提供全基因组数据的阵列对37例患者进行分析。与一般趋势相比,这些病例是由于5p缺失太小而不能解释延迟水平的所有病例加上与趋势匹配的几乎相同数量的病例。观察到严重延迟和轻微5 p缺失的受试者均观察到额外的增加和减少 (例如45名患者 [如图1C所示])。
对特定表型的影响不是由任何这些额外的异常引起的。发现异常的受试者的详细情况如を表4所示。由于这些测量使用具有进化克隆组成的阵列,因此定义附加像差边界的精度是可变的。然而,显而易见的是,这些异常通常包括至少一些连续的序列元素,并且明显大于非受累者的拷贝数多态性(Albertson和Pinkel 2003;Iafrate et al.2004;Sebatet al.2004)。
这些复杂例子中的一些包括影响第五染色体的其它异常,所述第五染色体包括位于缺失边界近端的一些重叠物质。 图2B示出了具有5p缺失且未检测到其他异常的所有患者的缺失与MR水平之间的关系。MR的严重程度与缺失的大小和位置之间的关系现在更加一致。
影响哭声,声音延迟和面部形态异常的染色体区域
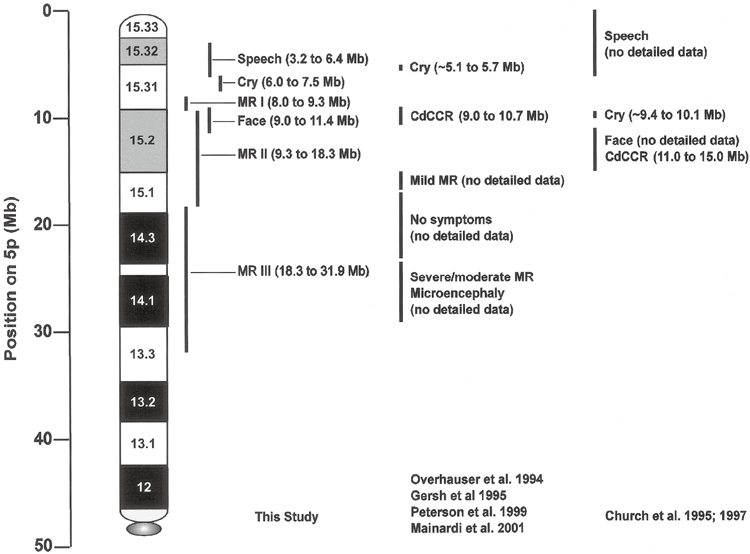
影响哭声、发声和面部特征的染色体区域先前映射到5p的远端部分(Overhauser et al.1994;Gersh et al.1995、1997)。
图3示出了具有缺失边界的受试者的数据,所述缺失边界提供关于这些基因型和表型之间关系的信息。在每种情况下,指定边界上的克隆体。影响特征性哭声的区域最窄地定义为患者49和252中的差异,两者都具有间质性缺失。对于患有cry的患者49而言,缺失可从克隆体12的近端开始,而对于未患有cry的患者252,缺失可保留至克隆体14的序列。因此,参与该表型的染色体区域位于这些克隆之间的1.5 Mb区域。
影响特征面部特征的区域由患者117、202和114定义得最窄。患者117具有在克隆体17和18之间开始的末端缺失,且不具有面部表型。因此,关键区域必须位于克隆体17的近端。这是因为材料可以保持到这个位置。患者202具有较大的末端缺失,在克隆体26和27之间具有边界,并且具有表型。这将克隆体17和27之间的2.4-Mb区域定义为负责面部特征的区域。患者114支持该区域的近端限制。患者114具有间质性缺失并且具有在克隆体26和27之间的远端边界,并且不具有表型,并且该表型的关键位置位于克隆体27的远端。本发明人注意到,具有包括所提出的临界区域的缺失的患者252是不一致的,因为患者不具有表型。
涉及言语延迟的染色体区域最好由102、225和49患有间质性缺失的患者的数据定义。在具有表型的患者102和225中,缺失接近克隆体4。在没有表型的患者49中,染色体的远端部分可以保留至克隆体13。因此,声音延迟是由于克隆体4和13之间的3.2-Mb区域中的缺失。
考察
建立发病综合征患者基因型和表型之间的关系是发现导致症状的遗传机制的第一步,并为临床管理患者疾病提供基础。许多关于导致猫叫综合症的染色体异常的先前研究已经建立了这种关系的概述,如图4所示。已经定义了染色体5p上的几个区域,这些区域分别影响经典表型的不同成分,其中1个5p 15.2对MR有很强的影响,被称为 「关键声音临界区域」 (CdCCR) ,其他与特征哭声,语言延迟,面部特征有关。然而,各种研究之间存在显着差异,包括MR和缺失之间关系的讨论。本研究使用阵列CGH对精心评估表型的患者组的DNA拷贝数变化进行了有效的高分辨率分析,提供了有关表型特定方面的更准确信息,并揭示了一些有争议的问题。
很难确定缺失大小与MR之间的关系,并且一些研究表明,随着缺失大小的增加,影响的严重性逐渐增加 (Mainardi等人在2001年没有发现这种关系(Marinescu et al.1999 a)。
在这项研究中,在测量 (Niebuhr1978a,1978b;Overhauser等) 之前的许多年,由单一观察者 (E.N.) 以一致的方式在数值尺度上评估MR状态分析了一系列患者。1990年, Kjaer和Niebuhr 1999年。
由于延迟的评估可能取决于患者的年龄,因此我们仅针对5岁或以上的患者进行评估。由于所有分析都使用从外周血分离的DNA而不是EB病毒转化细胞系,因此转化诱导的异常不会干扰我方的结果。
图2A表示5p的缺失与MR水平的关系。很明显, MR水平高达约3的患者的缺失小于MR水平5或更高的患者,但延迟对缺失大小的依赖性似乎较弱。例如,在MR水平5或更高时,由于不同延迟水平之间的缺失范围几乎完全重叠,因此具有-5级MR的许多患者具有与具有-7级MR的患者类似的5 p缺失。此外,还有一些明显的分歧。患者112和114具有约10 Mb的间质性缺失, MR水平为1~2;然而,严重受累的患者45在相同区域具有小得多的间质性缺失。
细胞遗传学分析先前表明,在一些猫叫综合症患者中,发现复杂的染色体重排,涉及基因组的其他区域的复制异常。因此,作者对37名患者进行了全基因组阵列CGH,包括所有因5p缺失太小而无法解释延迟水平的患者。如 表4所示,在这些患者中的15例中,除了5 p缺失之外还观察到异常。研究结果表明,大部分 (14/22) 明显延迟的受试者除5 p缺失外还有异常。如果将注意力转移到其中唯一检测到的异常缺失为5p的受试者,则延迟对缺失大小和位置的依赖性将更加明显。图2B示出了对于延迟水平3.5或更高,随着缺失大小的增加,延迟单调地增加。 这种与一般行为的轻微偏离可能是由于患者遗传因子的修饰、MR水平确定的不确定性或在基因组的其他部位未检测到的小异常。注意,如图2B所示, MR和缺失之间的明确关系支持患者表型评估的准确性。因为MR偶然发生的可能性极低。没有患者的缺失边界在5 pter到33 Mb的近端,这可能表明这种缺失在发展过程中是致命的。
与标准细胞遗传学相比,阵列CGH提供了更完整的基因组评估。总体而言,大约16%的受试者观察到复杂的异常,略大于先前测试的12%~13% (Niebuhr1978a;Mainardi et al.) 。2001) .
在阵列CGH (例,患者206)中观察到新的剂量异常的许多 (不是所有) 受试者中观察到细胞遗传学检测的结构异常,包括5号染色体和其他染色体(表1)。
相反,在具有染色体间细胞遗传学异常的一部分受试者(例如,患者4和49)中,除了染色体5p以外,未观察到复制数的变化。
因此,阵列CGH的较高分辨率和效率对于评估猫叫综合症中的剂量异常具有重要的优点,并且对于特别严重的延迟患者具有明显的诊断优点,其中约2/3具有中度5p缺失和额外的异常。
如图2B所示, MR依赖于5p缺失,这表明存在对该表型具有不同影响的三个染色体区域。初始MRI包括所有延迟水平为2.5或更高的患者的缺失。患者117和56的发现表明MRI可能局限于5p 15.31的克隆15和18之间的1.2-Mb区域,如其他文献中所定义,其位于5p 15.2的CdCCR末端(Overhauser et al.1994;Mainardi et al.2001)。
目前尚不清楚MRI中有多少重要基因,但由于其大小有限,因此表明可能很少。
如图2B所示,其他两个区域MRII和MRIII紧邻MRI。仅限于MRII的缺失具有轻度影响,而仅影响MRIII的缺失(例,患者51)不会产生可识别的表型。然而,具有包括MRI的缺失的患者随着缺失延伸到MRII和MRIII而逐渐严重延迟。因此, MRII和MRIII中的一系列基因似乎对表型有贡献,可能是通过修饰MRI中的基因效应。
当然,这些边界的定义取决于是否采用基础模型,该模型暴露于一些不确定性,并且缺失如何影响MR。基本上,本模型在MRI中起主导作用, MRII和MRIII中的进一步异常将显着增加表型的严重程度。MRI的远侧边界是由以下事实设定的:比克隆15更远的基因的缺失或保留对我们患者的MR水平没有明显影响。然而,在MRI的远端区域没有局部缺失的患者,并且不能绝对地确认。缺失可能表明由于缺乏MR表型而缺乏确认,但是这样的个体将期望具有猫叫综合症的其他特征。
建立MRI的近端边界更有问题。
以上,通过使用患者117的边界,示出了与数据一致的最小可能区域的选择。然而,在较不严重且具有较严重延迟水平的其他受试者中,观察到稍大的缺失,并且在克隆体26~28附近观察到切割点簇区域的边界。因此,患者117在某种程度上是不正常的,因此MRI可以相对于克隆体28进一步向远端延伸2Mb。为了更清楚地定义该区域,有必要分析具有充分表征的表型的许多附加病例。
仅限于MRII和MRIII的缺失导致轻度患病或表型正常的个体。然而,相对正常的表型似乎是「脆弱的」。因为其他异常导致严重的表型。
这些额外的异常包括如上所述的MRI的缺失和/或在第五染色体(对于患者25、48、104、201和251)或其他染色体(表4)上的其他部位的增加或减少。
例如,明显延迟的患者45在5p的MRII中仅有少量缺失,这将导致非常轻微的延迟。因此,6q的额外缺失 (图1C)可能导致非常严重的MR表型。注意到MRII和MRIII的增加可能会影响表型。在MR水平6.5的患者48缺少与延迟水平4~5一致的MRII和MRIII的一部分。这样, MRII和MRIII的一些重叠 (在缺失边界) 具有很强的效应。
家族数据还显示MRIII缺失个体的正常表型脆弱性。例如,患者51中的缺失存在于母亲和祖父母中,并且所有3个都不受影响。然而,有报道称患有MRIII缺失的未患病父母可能患有遗传缺失的患病儿童(Hand et al.2000, Johnson et al.2000)。
这种表型变化可能表明,对具有正常基因组的人没有有害影响的环境或遗传修饰因子将对具有MRIII缺失的人产生严重后果。或者,受影响的儿童可能具有在这些研究所采用的技术中未能检测到的进一步的异常。高分辨率全基因组阵列CGH可能检测到这种异常并发现这种儿童的基因型状态。
本研究还提供了染色体5p上区域的改善定位,这会影响特征性哭泣,面部特征和声音延迟。在cry区域中,通过仅缺失1个克隆体来区分具有表型和不具有表型的患者。这使得可能的关键基因位于两个侧翼克隆体 (分别包括标记物D5S2054和D5S676) 之间的阵列上。因此,对这两种情况的高分辨率分析将允许对临界区域设置更严格的极限值。因为多个阵列克隆体包含在其中,所以对当前病例的高误差消除分析不会使得语音延迟或面部区域变得非常精确。关于基因型-表型关系的研究结果与迄今为止发表的文献进行比较的结果如图4所示。
简而言之,为了更好地理解基因型和表型之间的关系,使用阵列CGH绘制了94例5p缺失患者的异常。作者指出,染色体的三个区域对患者的MR水平有不同的影响。涉及这三个区域的全部或部分的缺失与基因组中的其他异常相互作用以产生完整的MR表型。最后,作者的高分辨率数据使得在猫叫综合症的典型哭泣,面部特征和语言延迟中涉及到的基因的位置得以细化。
表2
MR分类
| MR水平 | 说明(IQ) | 表型 | |
|---|---|---|---|
| 儿童 | 幼儿 | ||
| 0 | 正常 | 没有MR | 没有MR |
| 1 | 界限 (<70) |
长期在正规学校学习,需要少量/主要的帮助 | 正常发育的里程碑,上学的第一年有明显的轻度延迟 |
| 2 | 非常稳定 (<65) |
上几年正规学校,需要很大的支援,有简单的读写和数学能力。 | 最初几年是正常发育的里程碑,2-3岁开始有明显的轻度延迟 |
| 3 | 稳定 (<50) |
能理解包括长文在内的一切,具有非常简单的读写和算数的能力。 | 成长的里程碑推迟了几个月。 |
| 4 | 中等程度 (<35) |
理解大多数事物,并使用小文章和大量签名。 | 发育的里程碑推迟了几个月。从1岁开始有明显的延迟 |
| 5 | 重度 (<20) |
理解简单的常用语和单词,会使用2~3个单词的句子和多个签名,会走路。 | 发育的里程碑推迟几个月到一年。MR在1岁前变得明显 |
| 6 | 非常严重 (<10) |
能理解几个单词。正常情况下,如果受到支持,就会走得很不稳定。不会说话,或者只会说几句话。 | 成长的里程碑已经晚了几年;MR在6个月前被发现 |
| 7 | 极度 | 症状轻微或无反应,可坐下或站立。 | 到5岁还不能一个人坐立。 |
表3
5p分析阵列克隆体
| 克隆数 | 标记物 | 位置 | 克隆名称 | |||
|---|---|---|---|---|---|---|
| 记号 | 名称 | 细胞遗传学a | 排列(Mb)a | 遗传的(cM)b | ||
| 1 | D5S1981 | AFMa217zh1 | 5p15.33 | 1.207 | 1 | CTC-326E20 |
| 2 | D5S2005 | AFMB002xc1 | 5p15.33 | 1.395 | 0 | RP11-94J21 |
| 3 | D5S1970 | AFMa183wh5 | 5p15.33 | 2.497 | 5 | CTB-116F8 |
| 4 | D5S417 | AFM205wh8 | 5p15.33 | 3.174 | 6 | RP11-20B3 |
| 5 | D5S1980 | AFMA217yh1 | 5p15.33 | 3.449 | 7 | RP11-82M24 |
| 6 | D5S675 | AFM336tc1 | 5p15.33 | 3.998 | 9 | RP11-103L11 |
| 7 | D5S1906 | WI-2725 | 5p15.33 | 4.258 | CTB-27O23 | |
| 8 | D5S405 | AFM154xa3 | 5p15.33 | 3.995 | 9 | RP11-227M19 |
| 9 | D5S406 | AFM154xg3 | 5p15.32 | 5.047 | 12 | RP11-58A5 |
| 10 | D5S1921 | WI-2897 | 5p15.32 | 5.200 | CTC-263B18 | |
| 11 | D5S464 | AFM112xe3 | 5p15.32 | 5.890 | 15 | CTC-248O4 |
| 12 | D5S2054 | AFMB355wb1 | 5p15.32 | 5.945 | RP11-53K22 | |
| 13 | D5S635 | AFM276yb9 | 5p15.32/15.31 | 6.365 | 16 | RP11-36H5 |
| 14 | D5S676 | AFM347yg9 | 5p15.31 | 7.492 | 18 | RP11-46O23 |
| 15 | D5S18c | 5p15.31 | 8.021 | CTB-16G3 | ||
| 16 | D5S1957 | AFMa124wg5 | 5p15.31 | 8.550 | 20 | CTB-27I9 |
| 17 | D5S208 | 5p15.31 | 9.039 | 21 | DPA-896A3 | |
| 18 | D5S74d | 5p15.31 | 9.288 | DPA-671G7 | ||
| 19 | D5S630 | AFM268zd9 | 5p15.31 | 9.614 | 21 | DPA-255H3 |
| 20 | D5S759 | AFM204ze1 | 5p15.31/15.2 | 10.100 | DPA-1398C5 | |
| 21 | D5S1850 | WI6722 | 5p15.2 | 10.328 | DPA-1104F2 | |
| 22 | D5S23e | 5p15.2 | 10.364 | DPA-1349G2 | ||
| 23 | AFM042xa11 | 5p15.2 | 10.403 | 23 | RP11-145B1 | |
| 24 | D5S2480 | WI7320 | 5p15.2 | 10.732 | DPA-941C2 | |
| 25 | D5S432 | AFM255xb9 | 5p15.2 | 10.746 | RP11-72C10 | |
| 26 | D5S117 | 5p15.2 | 11.360 | DPB-70G7 | ||
| 27 | D5S2887 | AFMa240xf9 | 5p15.2 | 11.435 | 25 | RP11-29N3 |
| 28 | D5S478
f |
AFM179xd10 | 5p15.2 | 11.035 | 24 | CTC-305H11 |
| 29 | D5S2081 | AFM347ta5 | 5p15.2 | 13.530 | 26 | CTC-305H17 |
| 30 | D5S1991 | AFMa282wa5 | 5p15.2/15.1 | 14.929 | 29 | RP11-5N8 |
| 31 | D5S1989 | AFMa247wc1 | 5p15.1 | 15.679 | 30 | RP11-135M13 |
| 32 | D5S1954 | AFMA114xf9 | 5p15.1 | 15.872 | 30 | RP11-261B20 |
| 33 | D5S1963 | AFMA140vd1 | 5p15.1 | 16.684 | 31 | RP11-269O14 |
| 34 | D5S416 | AFM205wh10 | 5p15.1 | 16.723 | 31 | RP11-260E18 |
| 35 | D5S2114 | AFMa090yh5 | 5p15.1 | 16.949 | 32 | CTB-28D11 |
| 36 | D5S486 | AFM206zc1 | 5p15.1 | 17.224 | 34 | CTB-33B3 |
| 37 | D5S2096 | AFM105xg1 | 5p15.1 | 17.500 | 35 | RP11-88L18 |
| 38 | WI-4804 | 5p15.1/14.3 | 18.336 | CTB-34B4 | ||
| 39 | D5S2419g | WI-10830 | 5p14.3 | 20.837 | CTC-253L1 | |
| 40 | D5S411 | AFM193xe11 | 5p14.3 | 21.797 | 39 | CTB-55P22 |
| 41 | D5S1868 | WI-9400 | 5p14.3/14.2 | 23.276 | CTB-115E13 | |
| 42 | D5S648 | AFM292yg5 | 5p14.1 | 25.841 | 42 | CTB-100A5 |
| 43 | D5S627 | AFM217ye1 | 5p14.1 | 26.794 | 43 | CTC-296N5 |
| 44 | D5S661/D5s2061 | AFMC011xb1 | 5p14/13 | 30.022 | 44 | CTB-161H9 |
| 45 | D5S477f | AFM177xb4 | 5p13.3 | 31.942 | 49 | |
| 46 | D5S1993 | AFMa286ya9 | 5p13.3 | 31.751 | 48 | CTB-13O21 |
| 47 | D5S1986 | AFMA238za9 | 5p13.3 | 31.776 | 49 | RP11-5N11 |
| 48 | D5S1996 | AFM297wa5 | 5p13.3 | 32.162 | 51 | RP11-67P13 |
| 49 | D5S651 | AFM302wd5 | 5p13.3 | 33.428 | 51 | CTC-221E3 |
| 50 | D5S2062 | AFM277yh9 | 5p13.3 | 33.806 | 55 | RP11-94E6 |
| 51 | D5S395f | AFM284vc1 | 5p13.3 | 30.726 | CTB-62F24 | |
| 52 | D5S1506 | GATA63c02 | 5p13.3 | 33.873 | CTB-38G24 | |
| 53 | D5S395 | AFM063yb6 | 5p13.2 | 35.843 | 57 | RP11-85N3 |
| 54 | D5S2025 | AFMb297za5 | 5p13.2 | 36.018 | 57 | CTB-107L17 |
| 55 | D5S1994 | AFMa286ze9 | 5p13.2 | 36.465 | 56 | CTB-18O17 |
表4 额外异常
| 患者 | MR 水平 |
细胞遗传学位置 | 复制数量 | 上限 | 下限 | 大小 (Mb) |
修订核型a | ||
|---|---|---|---|---|---|---|---|---|---|
| 保留克隆体b | 位置 (Mb)c |
保留克隆体b | 位置 (Mb)c |
||||||
| 16 | 6.5 | 8pter-21.3 | 3 | pter | 0 | RP11-89M8 | 22.745 | 22.745 | 46,XX,der(5)t(5;8)(p14.1;p21) |
| 24 | 7 | 13q32.2-ter | 3 | RP11-40H10 | 93.26 | qter | 113.042 | 19.782 | 46,XX,der(5)t(5;13)(p14.1;q32.2) |
| 25 | 7 | 5q35.3-ter | 3 | RP11-125L2 | 173.496 | qter | 181.034 | 7.538 | 46,XX,der(5)t(5;5)(p14.2;q35.3)mat |
| 35 | 6.5 | 14q11.2 | 1 | RP11-152G22 | 19.227 | RP11-68M15 | 21.467 | 2.24 | 45,XY,der(5)t(5;14)(p14.2;q11.2), -14,mat |
| 45 | 7 | 6q25.2-6qter | 1 | GS-59B4 | 152.455 | RP1-57H24 | 170.695 | 18.24 | 46,XY,der(14)t(5;14)(q11;p13) del(5)(p15.1p15.2)del(6)(q25.2) |
| 48 | 6.5 | 5p15.11-5p14 | 3 | RP11-261B20/32d | 15.872 | CTC-296N5/43 | 26.794 | 10.922 | 46,XY,del(5)(p14.3)dup(5)(p14.2p15.1) |
| 104 | 6.5 | 5p13.3 | 4 | CTB-13O21/46 | 31.715 | RP11-67P13/48 | 32.162 | .447 | 46,XY,del(5)(p13.3)dup(5)(~p13.3)e×2 |
| 201 | 7 | 5q35.1-terf | 3 | RP11-15F10 | 169.122 | qter | 181.034 | 11.912 | 46,XY,der(5)t(5;5)(p14.3q35.1) |
| 206 | 7 | 20pter-12.2 | 3 | pter | 0 | DuPontA-967A8 | 10.282 | 10.282 | 46,XX,der(5)t(5;20)(p14.3;p12.2)pat |
| 210 | 7 | 1qter | 3 | RP11-194F13 | 238.741 | qter | 246.127 | 7.386 | 46,XX,der(5)t(1;5)(q43;p14.1)pat |
| 215 | 7 | 4q31.1-ter | 3 | RP11-40D5 | 135.249 | qter | 191.731 | 56.482 | 46,XX,der(5)t(4;5)( q31.1;p14.3) |
| 218 | 6.5 | 8pter-8p23.2 | 3 | pter | 0 | RP11-112O08 | 11 | 11 | 46,XX,der(5)t(5;8)(p14.3;p23.2)pat |
| 228 | 6 | 8q23.1-ter | 3 | CTD-2013D21 | 110.377 | qter | 146.308 | 35.931 | 46,XY,der(5)t(5;8)(p15.2;q23.1)pat |
| 251 | 6.5 | 5p13.3 | 3 | CTB-115E13/41 | 23.276 | RP11-94E6/50 | 33.806 | 10.53 | 46,XY,del(5)(p13.3)dup(5)(p13.3~14.1)e |
| 255 | 6.5 | 1qter | 3 | RP11-210E16g | 229.578 | 46,XY,der(5)t(1;5)(q42~43;15.2) | |||
图2
MR水平对5p缺失的依赖性。A、延迟评估可用的全部91例数据。黑色部分表示在每种情况下保留的染色体区域,并相对于5pter的物理位置绘制。中心位于图表底部下方。延迟水平对缺失的依赖性是显而易见的,但往往伴随着在一般趋势中看起来太小的缺失。对于MR水平5,按照每个水平内的近端缺失边界的顺序绘制数据。
从这些重症和重度患病患者分配延迟表型的MR水平估计有0.5~1的不确定性。对于仅检测到5p缺失的患者,也采用了B,相同的绘图。需要注意的是,大多数严重障碍的患者并不存在,因为存在更多的异常。延迟对5p缺失的依赖性要一致得多。建议5p-MRI、MRII和MRIII三个区域对延迟具有差异效应。本文将对这些领域进行说明。
图3 Am. J. Hum. Genet. 76:312–326, 2005
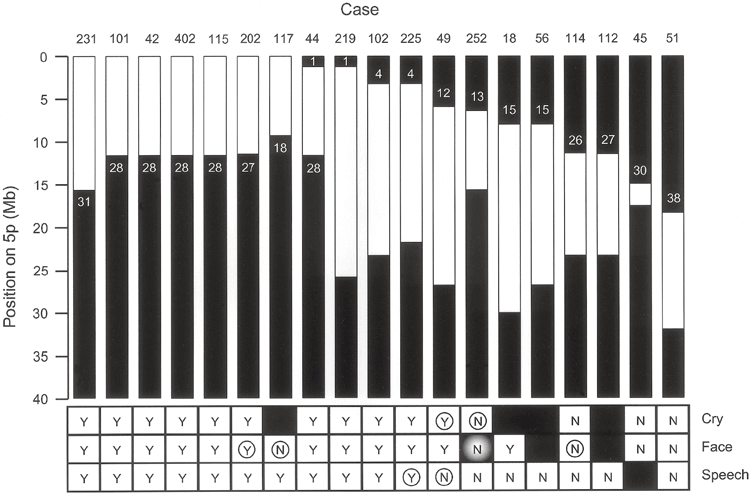
黑色方块表示无法获得与该患者相关的信息。圆圈表示患者提供关于引起该特征的染色体部分的位置的最有用信息。患者252的网状「N」 表示该患者的缺失应不会导致表型。
图4 Zhang et al.: Cri du Chat Genotype-Phenotype
感谢
该研究得到了国家儿童卫生与人类发展研究所资助HD 17665, VISIS有限公司,丹麦STF比较基因组计划,丹麦基础研究基金会综合生物学平台,国家自然科学基金会的赞助。
电子数据库信息
- 此处发布的数据的URL如下。在线孟德尔遗传 (OMIM) , http://www.ncbi.nlm.nih.gov/Omim/, UCSC基因组生物信息学, http://genome.cse.ucsc.edu/参考文献Albertson DG, Pinkel D (2003) 人类遗传病和癌症的基因组微阵列。
参考文献
- Hum Mol Genet 12 (Spec 2) :通过R 145 -152 Albertson DG, Ylstra B, Segraves R, Collins C, Darkee SH, Kowbel D, Kuo WL, Gray JW, Pinkel D (2000) 阵列CGH定量映射扩增子结构,证实CYP 24是候补癌基因。
- Nat Genet 25:144 -146 Church DM, Bengtsson U, Nielsen KV, Wasmuth JJ, Niebuhr E (1995) 远端5p的不同片段缺失的分子定义导致不同的表型特征。
- Am J Hum Genet 56:1162 -1172 Church DM, Yang J, Bocian M, Shiang R, Wasmuth JJ (1997) 人类染色体5p猫叫区域的高分辨率物理和转录图。
- Genome Res 7:787 -801 Eichler EE (2001) 最近的重复,域名的添加和人类基因组的动态突变。
- Trends Genet 17:661 -669采用Fiegler H, Carr P, Douglas EJ, Burford DC, Hunt S, Scott CE, Smith J, Vetrie D, Gorman P, Tomlinson IP, Carter NP (2003) DNA微阵列进行基于BAC和PAC克隆DOP-PCR扩增的比较基因组混合。
- Genes Chromosomes Cancer 36:361-374Gersh M, Goodart SA, Pasztor LM, Harris DJ, Weiss L, Overhauser J (1995) 5p缺失的患者中导致猫叫声的不同区域的证据。
- Am J Hum Genet 56:1404 -1410使用Gersh M, Grady D, Rojas K, Lovett M, Meyzis R, Overhauser J (1997) 期间FISH分析5p缺失开发诊断工具
- Cytogenet Cell Genet 77:246 -251 Hand JL, Michels VV, Marinello MJ, Ketterling RP, Jalal SM (2000) 染色体5p和16q的间质性缺失遗传无明显的表型效应:furth确认。
- Prenat Diagn 20:144-148 Hudson TJ、Stein LD、Gerety SS、Ma J、Castle AB、 Silva J、 Silva J、 Slonim DK、Baptista R、Kruglyak L、Xu S-H、Hu X、Colbert AME、Rosenberg C、Reeve-Daly MP、Rozen S、Hui L、Wu X、Vestergaard C、Wilson KM、Bae JS、Maitra S、Ganiatsas S、Evans CA、DeAngelis MM、Ingals KA(1995) 基于STS的人类基因组图谱。
- Science 270:1945-1954 Iafrate AJ, Feuk L, Rivera MN, Listewnik ML, Donahoe PK, Qi Y, Scherer SW, Lee C (2004) 检测人类基因组中的大规模突变
- Nat Genet 36:949-951 Jain AN, Tokuyasu TA, Snijders AM, Segraves R, Albertson DG, Pinkel D (2002) 微阵列图像数据的全自动量化
- Genome Res 12:325-332 Johnson EI, Marinescu RC, Punnett HH, Tenenholz B, Overhauser J (2000) 5p14缺失与小头畸形和癫痫发作有关。
- J Med Genet 37:125 -127 Kjaer I, Niebuhr E (1999) 对23例猫叫综合症患者颅内底部的研究表明,这种情况涉及颅骨发育。
- Am J Med Genet 82:6-14 Levy B, Dunn TM, Kern JH, Hirschhorn K, Kardon NB (2002) dup5q/del 5p(cri du chat)通过患者分子细胞遗传学分析的dup5q表型Delineation
- Am J Med Genet 108:192-197 Lupski JR, Roth JR, Weinstock GM (1996) 细菌,果蝇和人类的染色体重叠。
- Am J Hum Genet 58:21-27 Mainardi PC, Perfumo C, Cali A, Coucourde G, Pastore G, Cavani S, Zara F, Overhauser J, Pierluigi M, Bricarelli FD (2001)80例5p缺失患者的临床和分子特征:基因型-表型相关性
- J Med Genet 38:151-158 Marinescu RC, Johnson EI, Dykens EM, Hodapp RM, Overhauser J (1999a) 缺失的大小与猫叫综合症中发育延迟的水平无关。
- Am J Med Genet 86:66-70 Marinescu RC, Johnson EI, Grady D, Chen XN, Overhauser J (1999b) 诊断为猫叫综合症患者末端缺失的FISH分析
- Clin Genet 56:282 -288 Niebuhr E (1978 a) 5p-核型35例的细胞学观察
- Hum Genet 42:143 -156— (1978 b) 猫叫综合症:流行病学,细胞遗传学和临床特征。
- Hum Genet 44:227 -275 Overhauser J, Huang X, Gersh M, Wilson W, McMahon J, Bengtsson U, Rojas K, Meyer M, Wasmuth JJ (1994) 第5号染色体短臂的分子和表型图谱:猫叫综合症临界区域的亚定位。
- Hum Mol Genet 3:247 -252 Overhauser J, McMahon J, Oberlender S, Carlin ME, Niebuhr E, Wasmuth JJ,Le-ChenJ (1990) 猫叫综合症中染色体5缺失的父母来源。
- Am J Med Genet 37:83 -86 Peterson ET, Sutherland R, Robinson DL, Chasteen L, Gersh M, Overhauser J, Deaven LL, Meyzis RK, Grady DL (1999) 人类5号染色体短臂的综合物理地图。
- Genome Res 9:1250-1267 Pinkel D, Segraves R, Sudar D, Clark S, Poole I, Kowbel D, Collins C, Kuo WL, Chen C, Zhai Y, Dirkee SH, Ljung BM, Gray JW, Albertson DG (1998) 使用基因组混合进行DNA拷贝数突变的高分辨率分析
- Nat Genet 20:207使用-211 Pollack JR, Perou CM, Alizadeh AA, Eisen MB, Pergamenschikov A, Williams CF, Jeffrey SS, Bo stein D, Brown PO (1999) cDNA微阵列进行DNA拷贝数变化的全基因组分析
- Nat Genet 23:41 -46 Sebat J, Lakshmi B, Troge J, Alexander J, Young J, Lundin P, Maner S, Massa H, Walker M, Chi M, Navin N, Lucito R, Healy J, Hicks J, Ye K, Reiner A, Gilliam TC, Trask B, Patterson N, Zetterberg A, Wigler M (2004) 人类基因组中的大规模拷贝数多态性。
- Science 305:525-528Snijders AM、Nowak N、Segraves R、Blackwood S、Brown N、Conroy J、Hamilton G、Hindle AK、Huey B、Kimura K、Law S、Myambo K、Palmer J、Ylstra B、Yue JP、Gray JW、Jain AN、Pinkel D、Albertson DG (2001) 组装用于全基因组DNA拷贝数测量的微阵列
- Nat Genet 29:263 -264Solinas-ToldoS, Lampel S, Stilgenbauer S, Nickolenko J, Benner A, Dohner H, Cremer T, Lichter P (1997) 基于矩阵的比较基因组混合:筛选基因组失衡的生物芯片。
- Genes Chromosomes Cancer 20:399 -407 Sreekantaiah C, Kronn D, Marinescu RC, Goldin B, Overhauser J (1999) 具有典型的猫一样的哭声并且没有其他猫叫综合症临床表现的患者的复杂染色体重组的特征。
- Am J Med Genet 86:264 -268 Wilkins LE, Brown JA, Nance WE, Wolf B (1983) 80例猫叫综合症患儿的临床异质性
- J Pediatr 102:528-533
出版商注释
Springer Nature对公开的地图和设施内的合作中的管辖权主张是中立的。
 中文
中文